La Revolución de las Proteínas: Simulando 500 Millones de Años de Evolución
Un Premio Nobel que Marca el Camino
El pasado mes de octubre, el comité del Premio Nobel en química otorgó este prestigioso galardón a David Baker por su trabajo en el diseño computacional de proteínas, y también a Demis Hassabis y John M. Jumper por sus innovaciones en la predicción de estructuras proteicas. Estos logros han abierto nuevas puertas en el estudio de proteínas, impulsando investigaciones en diversos laboratorios alrededor del mundo.
Nueva Vida en el Laboratorio
Un ejemplo destacado es la empresa EvolutionaryScale, que ha logrado crear una proteína fluorescente artificial a través de un modelo de inteligencia artificial. En sus propias palabras, esta creación representa la simulación de 500 millones de años de evolución natural, marcando un avance significativo en la biotecnología.
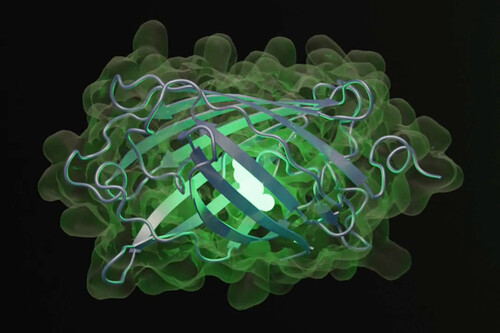
GFP: Las Proteínas Fluorescentes Verdes
La nueva proteína, conocida como esmGFP, pertenece a la familia de las proteínas fluorescentes verdes, o GFP (Green Fluorescent Protein). Este tipo de proteínas se encuentra en la naturaleza, como las que se encuentran en ciertas medusas, y su descubrimiento le valió a sus descubridores un Premio Nobel en 2008. Aunque la esmGFP comparte similitudes con las GFP existentes, su estructura y forma son diferentes, lo que la hace única y fascinante.
Evolución Simulada: Imaginando Nuevas Realidades
A pesar de que no se ha encontrado evidencia de que esta proteína artificial exista en la naturaleza, su funcionalidad permite a los científicos especular sobre cómo podría haber evolucionado la vida bajo diferentes circunstancias. Las diferencias observadas entre la esmGFP y otras proteínas pueden ser comparables a 500 millones de años de evolución natural, lo que representa una cantidad sorprendente de tiempo en el ámbito biológico.
Concepción de ESM3: Un Modelo Innovador
El equipo de EvolutionaryScale desarrolló un modelo generativo de lenguaje, conocido como ESM3 (EvolutionaryScale Model 3). A pesar de su nombre, este modelo no genera texto, sino que produce proteínas. Gracias a ESM3, se pueden estudiar infinitas combinaciones de secuencias y estructuras, lo que enriquese nuestras herramientas de investigación en biología y medicina.
Entrenamiento y Datos a Gran Escala
Para entrenar el modelo, se utilizaron 771.000 millones de paquetes de datos derivados de 3.150 millones de secuencias de proteínas, 236 millones de estructuras y 539 millones de proteínas con funciones relacionadas. Estos resultados se publicaron en un artículo en la prestigiosa revista Science, destacando la importancia del trabajo del equipo de EvolutionaryScale.
Más Allá de la Ficción: Aplicaciones Prácticas
El desarrollo de estas proteínas hipotéticas no solo nos permite explorar «qué podría haber sido», sino que también tiene implicaciones prácticas muy relevantes. Una de las aplicaciones más prometedoras es en el campo de la medicina, donde encontrar nuevas proteínas con funciones similares a las que produce el cuerpo humano puede ser crucial en la lucha contra diversas enfermedades y trastornos.
Conclusiones
La creación de proteínas artificiales, como la esmGFP, abre un mundo de posibilidades en la biología moderna. Nos invita a reflexionar sobre cómo la inteligencia artificial y la biotecnología pueden trabajar juntas para descubrir nuevos caminos en la evolución de la vida y en la mejora de la salud humana.